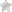Was wir hier sehen ist ein tragbarer Oxford Nanopore-DNS-Sequenzierer in Aktion mit einer Software namens UNCALLED, angeschlossen über einen USB-Port eines Laptops:

Das Revolutionäre auf diesem Foto ist nicht mal das portable Gerät, das ist als Gizmo alleine schon ziemlich modern und cool.
Sondern die UNCALLED-Software, ein OpenSource-Kunststück: ermöglicht sie nämlich die Bestimmung von DNS, also dem Quellcode von Leben, in Rekordzeit – sie wird verkürzt auf 3 Tage statt 15, manchmal reicht sogar schon ein einziger Tag. [via]
Funktioniert so: man braucht ein DNS-Sample, beispielsweise Blut oder ein bisschen Speichel. Dieses wird in Milliarden, manchmal Billionen kleinere Teile aufgeteilt, sogenannte fragmentierte Moleküle (schätze das sind die „Nanopores“, es gibt keinen deutschsprachigen Wikipedia-Artikel dafür…). Über eine Pipette (siehe Bild) fügt man anschließend die Lösung dem Nanopore-USB-Sequenzierer zu. Dieses Gerät kostet nur wenige Tausend Euro. Ein Sequenzierer nimmt nun diese Moleküle, das können schon einmal 512 oder mehr sein, und liest diese komplett ein, eins nach dem anderen. Dieser Vorgang war bisher sehr zeitaufwendig und wenig clever oder effizient.
Hier kommt nun UNCALLED ins Spiel: es prüft vorab und sehr schnell ob ein bestimmtes Molekül interessant für eine weitere Untersuchung ist. Wenn ja, wird es komplett ausgelesen wie bisher. Wenn nicht, stoppt die Software und ignoriert das Molekül, es wird ejected.
Die Software kann man sich als eine Art Tuner für die Nanopore-Hardware vorstellen: sie holt einfach Effizienz und Schnelligkeit aus dem vorhandenen Gerät raus. Etwas, was Hacker generell sehr oft machen, wenn sie an Software-Code arbeiten – früher waren Hardwareverfügbarkeit und -leistung noch sehr begrenzt, man musste sich Gedanken machen wie man mit Code das Beste aus dieser herausholt. Videospiele auf Konsolen zu entwickeln mag man hier als gutes Beispiel herannehmen – oft ist die Hardware bereits viele Jahre alt, bis ein Studio einen Blockbuster-Titel entwickelt hat und veröffentlicht.
Ein praktisches Beispiel für die oben erwähnte Software gibt es zum Abschluss: in einem 3-Tages-Testlauf zur Untersuchung spezifischer Krebs-Gene wurden gleich viele relevante Ergebnisse zurückgeliefert wie durch fünf gleichzeitig laufende Untersuchungen. Die Forscher betonen, 3 Tage war beliebig gewählt – es könnte in einem abgeschlossen sein.
In der Praxis wird durch diese Methodik es bereits in naher Zukunft einfacher unbekanntes Erbgut zu untersuchen. Ein Beispiel: den Niederlanden war es erst möglich am 27. Februar den ersten COVID-19-Fall zu untersuchen, da war das gefährliche Virus bereits fast zwei volle Monate überall in der Welt. Fertig analysiert war es hingegen erst am 29. Februar. Ohne die Details zu kennen ist jedoch keine zuverlässige Testung möglich. Was das erfolgreiche Erkennen von SARS-CoV-2 bei Patienten in Holland bis in den März 2020 verschob. Verlorene und wertvolle Zeit, die in dem Falle sogar Menschenleben wert war.